Comment améliorer l'outil CRISPR pour l'édition de gènes?
07-08-2018
Parmi les avancées scientifiques les plus importantes de ces dernières années, citons la découverte et le développement de nouvelles méthodes de modification génétique des êtres vivants grâce à une technologie rapide et abordable appelée CRISPR. Les scientifiques de l’Université du Texas à Austin affirment maintenant qu’ils ont identifié une mise à niveau facile de cette technologie qui conduirait à une édition plus précise des gènes avec une sécurité accrue qui pourrait ouvrir la voie à une édition génétiquement sûre.
L'équipe de biologistes moléculaires a trouvé des preuves concluantes que Cas9, l'enzyme la plus populaire actuellement utilisée dans l'édition de gènes CRISPR et la première découverte, est moins efficace et moins précise que l'une des protéines CRISPR moins utilisées, appelée Cas12a.
Parce que Cas9 est plus susceptible d'éditer la mauvaise partie du génome d'une plante ou d'un animal, perturbant les fonctions saines, les scientifiques affirment que le passage au Cas12a conduirait à une édition plus sûre et plus efficace dans leur étude publiée le 2 août dans la revue Molecular Cell
L'objectif global est de trouver la meilleure enzyme que la nature nous a donnée et de la rendre encore meilleure, plutôt que de prendre la première découverte par accident historique », a déclaré Ilya Finkelstein, professeur adjoint de biosciences moléculaires et co-auteur de l'étude.
Les scientifiques utilisent déjà CRISPR, un mécanisme naturel utilisé par les bactéries pour se défendre contre les virus, pour en savoir plus sur les gènes humains, modifier génétiquement les plantes et les animaux et développer des avancées inspirées par la science fiction. au lard plus maigre. Beaucoup s'attendent à ce que le CRISPR mène à de nouveaux traitements pour les maladies humaines et à des cultures plus productives ou résistantes aux sécheresses et aux ravageurs.
Mais les systèmes CRISPR trouvés dans la nature ciblent parfois le mauvais endroit dans un génome, qui - appliqué aux humains - pourrait être désastreux, par exemple, ne pas corriger une maladie génétique et transformer des cellules saines en cellules cancéreuses.
Certaines études antérieures ont laissé entendre que Cas12a était plus sélectif que Cas9, mais les recherches précédentes étaient peu concluantes. Selon les chercheurs, cette dernière montre que Cas12a est un scalpel d'édition de gènes plus précis que Cas9 et ils expliquent pourquoi.
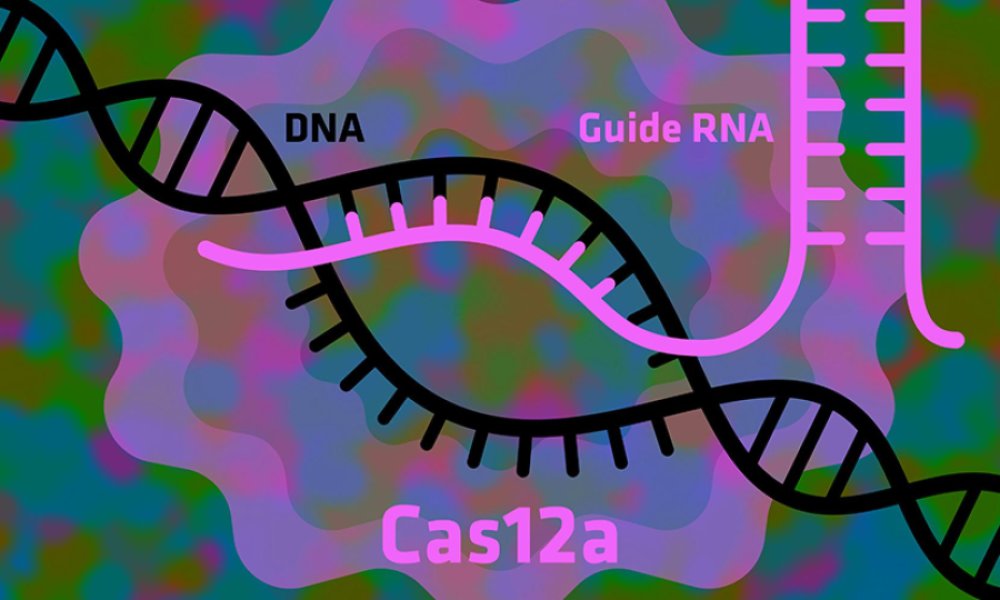
L’équipe, dirigée par l’étudiante Isabel Strohkendl et le professeur Rick Russell, a constaté que Cas12a est plus sélectif car il se lie comme une bande de Velcro à une cible génomique, alors que Cas9 se lie plus à sa cible comme une super colle. Chaque enzyme porte une courte chaîne de code génétique écrite en ARN qui correspond à une chaîne cible de code génétique écrite dans l'ADN d'un virus. Lorsqu'elle se heurte à un peu d'ADN, l'enzyme commence à essayer de s'y lier en formant des paires de bases - en commençant par une extrémité et en poursuivant son chemin, en vérifiant si chaque lettre d'un côté (l'ADN) correspond à la lettre adjacente de l'autre côté (l'ARN).
Pour Cas9, chaque paire de base adhère étroitement, comme avec une super colle. Si les premières lettres de chaque côté correspondent bien, alors Cas9 est déjà fortement lié à l'ADN. En d’autres termes, Cas9 prête attention aux sept ou huit premières lettres de la cible génomique, mais accorde moins d’attention au fur et à mesure que le processus se poursuit, ce qui signifie qu'il peut facilement ignorer une inadéquation ultérieure dans le processus qui l'amènerait à modifier la mauvaise partie du génome.
Pour Cas12a, cela ressemble plus à une bande velcro. À chaque étape du processus, les connections sont relativement faibles. Il faut un bon match tout au long de la bande pour que les deux parties se tiennent ensemble suffisamment longtemps pour effectuer un montage. Cela rend beaucoup plus probable qu'il ne modifiera que la partie du génome prévue .
Cela rend le processus de formation de paires de bases plus réversible », a déclaré Russell.« En d’autres termes, Cas12a vérifie mieux chaque paire de bases avant de passer à la suivante. Après sept ou huit lettres, Cas9 arrête de vérifier, alors que Cas12a continue à vérifier environ 18 lettres.
Les chercheurs ont déclaré que Cas12a n'est toujours pas parfait, mais l'étude suggère également des façons d'améliorer encore Cas12a, peut-être un jour en réalisant le rêve de créer un "scalpel de précision", un outil essentiel d'édition de gènes résistant aux erreurs.
Dans l’ensemble, Cas12a est meilleur, mais il y avait certaines zones où Cas12a était encore étonnamment aveugle à une mauvaise relation entre son ARN et la cible génomique », a déclaré Finkelstein.« Notre travail montre donc clairement la voie à suivre pour améliorer encore Cas12a . »
Les chercheurs utilisent actuellement ces informations dans un projet de suivi conçu pour concevoir un Cas12a amélioré.
Les autres co-auteurs de l'étude sont l'étudiant diplômé James Rybarski et l'ancienne étudiante de premier cycle Fatema Saifuddin.
Ce travail a été soutenu par des subventions du National Institute of General Medical Sciences et the Welch Foundation.
Traduction : Christina Lambrecht
Source : ScienceDaily
31-10-2017 - Un nouveau CRISPR Technique élimine C9orf72 ARN
13-10-2017 - L'équipe de l'Institut Parkinson utilise la saisie CRISPR / Cas9 et le séquençage PacBio
05-09-2017 - Le CRISPR-Cas9 pour modifier l’embryon humain
13-08-2017 - CRISPR ciblant l'ARN pourrait produire des traitements pour la maladie de Huntington et la SLA


